公司网站建设 公司简介怎么写网站中用特殊字体
计算机程序的构造和解释
我找到计算机里的神灵了,开心一刻
下面是从MIT官网下载的 SCIP求值器(解释器)的代码,这个官网是个宝藏库
还有其他视频课程和 SCIP的问题答案和可运行代码
链接:https://ocw.mit.edu/courses/6-001-structure-and-interpretation-of-computer-programs-spring-2005/pages/projects/
;;
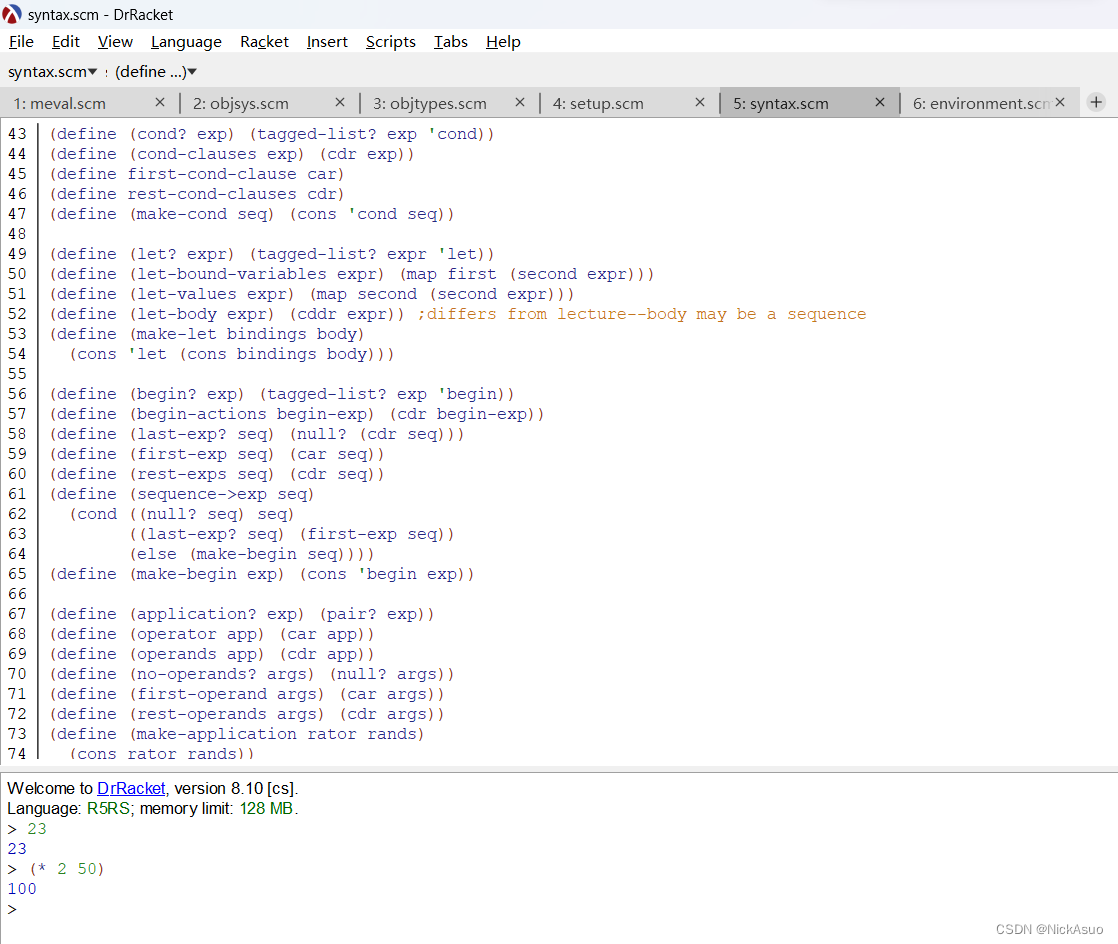
;; syntax.scm - 6.001 Spring 2005
;; selectors and constructors for scheme expressions
;;(define (tagged-list? exp tag)(and (pair? exp) (eq? (car exp) tag)))(define (self-evaluating? exp)(or (number? exp) (string? exp) (boolean? exp)))(define (quoted? exp) (tagged-list? exp 'quote))
(define (text-of-quotation exp) (cadr exp))(define (variable? exp) (symbol? exp))
(define (assignment? exp) (tagged-list? exp 'set!))
(define (assignment-variable exp) (cadr exp))
(define (assignment-value exp) (caddr exp))
(define (make-assignment var expr)(list 'set! var expr))(define (definition? exp) (tagged-list? exp 'define))
(define (definition-variable exp)(if (symbol? (cadr exp)) (cadr exp) (caadr exp)))
(define (definition-value exp)(if (symbol? (cadr exp))(caddr exp)(make-lambda (cdadr exp) (cddr exp)))) ; formal params, body
(define (make-define var expr)(list 'define var expr))(define (lambda? exp) (tagged-list? exp 'lambda))
(define (lambda-parameters lambda-exp) (cadr lambda-exp))
(define (lambda-body lambda-exp) (cddr lambda-exp))
(define (make-lambda parms body) (cons 'lambda (cons parms body)))(define (if? exp) (tagged-list? exp 'if))
(define (if-predicate exp) (cadr exp))
(define (if-consequent exp) (caddr exp))
(define (if-alternative exp) (cadddr exp))
(define (make-if pred conseq alt) (list 'if pred conseq alt))(define (cond? exp) (tagged-list? exp 'cond))
(define (cond-clauses exp) (cdr exp))
(define first-cond-clause car)
(define rest-cond-clauses cdr)
(define (make-cond seq) (cons 'cond seq))(define (let? expr) (tagged-list? expr 'let))
(define (let-bound-variables expr) (map first (second expr)))
(define (let-values expr) (map second (second expr)))
(define (let-body expr) (cddr expr)) ;differs from lecture--body may be a sequence
(define (make-let bindings body)(cons 'let (cons bindings body)))(define (begin? exp) (tagged-list? exp 'begin))
(define (begin-actions begin-exp) (cdr begin-exp))
(define (last-exp? seq) (null? (cdr seq)))
(define (first-exp seq) (car seq))
(define (rest-exps seq) (cdr seq))
(define (sequence->exp seq)(cond ((null? seq) seq)((last-exp? seq) (first-exp seq))(else (make-begin seq))))
(define (make-begin exp) (cons 'begin exp))(define (application? exp) (pair? exp))
(define (operator app) (car app))
(define (operands app) (cdr app))
(define (no-operands? args) (null? args))
(define (first-operand args) (car args))
(define (rest-operands args) (cdr args))
(define (make-application rator rands)(cons rator rands))(define (and? expr) (tagged-list? expr 'and))
(define and-exprs cdr)
(define (make-and exprs) (cons 'and exprs))
(define (or? expr) (tagged-list? expr 'or))
(define or-exprs cdr)
(define (make-or exprs) (cons 'or exprs))lisp解释器,就这样手撕出来了,看到神灵,万物跪拜