书籍教你如何做网站百度人工电话多少号
写在前面
Hello大家好, 我是【麟-小白】,一位软件工程专业的学生,喜好计算机知识。希望大家能够一起学习进步呀!本人是一名在读大学生,专业水平有限,如发现错误或不足之处,请多多指正!谢谢大家!!!
如果小哥哥小姐姐们对我的文章感兴趣,请不要吝啬你们的小手,多多点赞加关注呀!❤❤❤ 爱你们!!!
目录
写在前面
1. 图形化界面工具
1.1 概述
1.2 安装
1.3 使用
2. DML
2.1 添加数据
2.2 修改数据
2.3 删除数据
结语
【往期回顾】
【MySQL系列】 第一章 · MySQL概述
【MySQL系列】第二章 · SQL(上)
【其他系列】
【HTML5系列】
【HTML4系列】
【CSS2系列】
【CSS3系列】
【Java基础系列】
1. 图形化界面工具
1.1 概述
- 上述,我们已经讲解了通过DDL语句,如何操作数据库、操作表、操作表中的字段,而通过DDL语句执行在命令进行操作,主要存在以下两点问题:
- 会影响开发效率 ;
- 使用起来,并不直观,并不方便 ;
- 所以呢,我们在日常的开发中,会借助于MySQL的图形化界面,来简化开发,提高开发效率。而目前mysql主流的图形化界面工具,有以下几种:

- 我们使用最后一种DataGrip,这种图形化界面工具,功能更加强大,界面提示更加友好,是我们使用MySQL的不二之选。接下来,我们来介绍一下DataGrip该如何安装、使用。
1.2 安装
- 找到资料中准备好的安装包,双击开始安装
- 点击next,一步一步的完成安装
- 选择DataGrip的安装目录,然后选择下一步
- 下一步,执行安装

1.3 使用
添加数据源
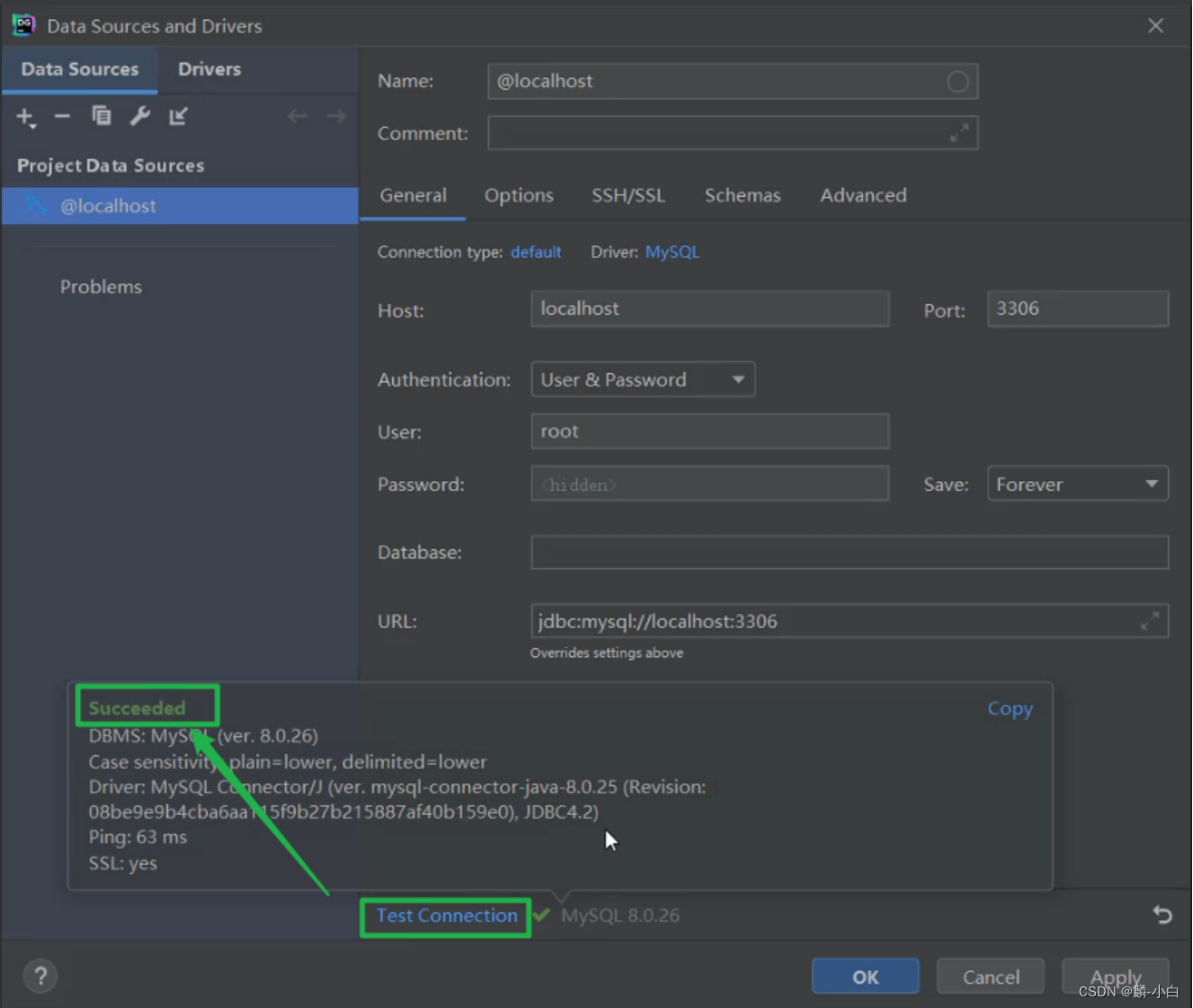
- 参考图示, 一步步操作即可
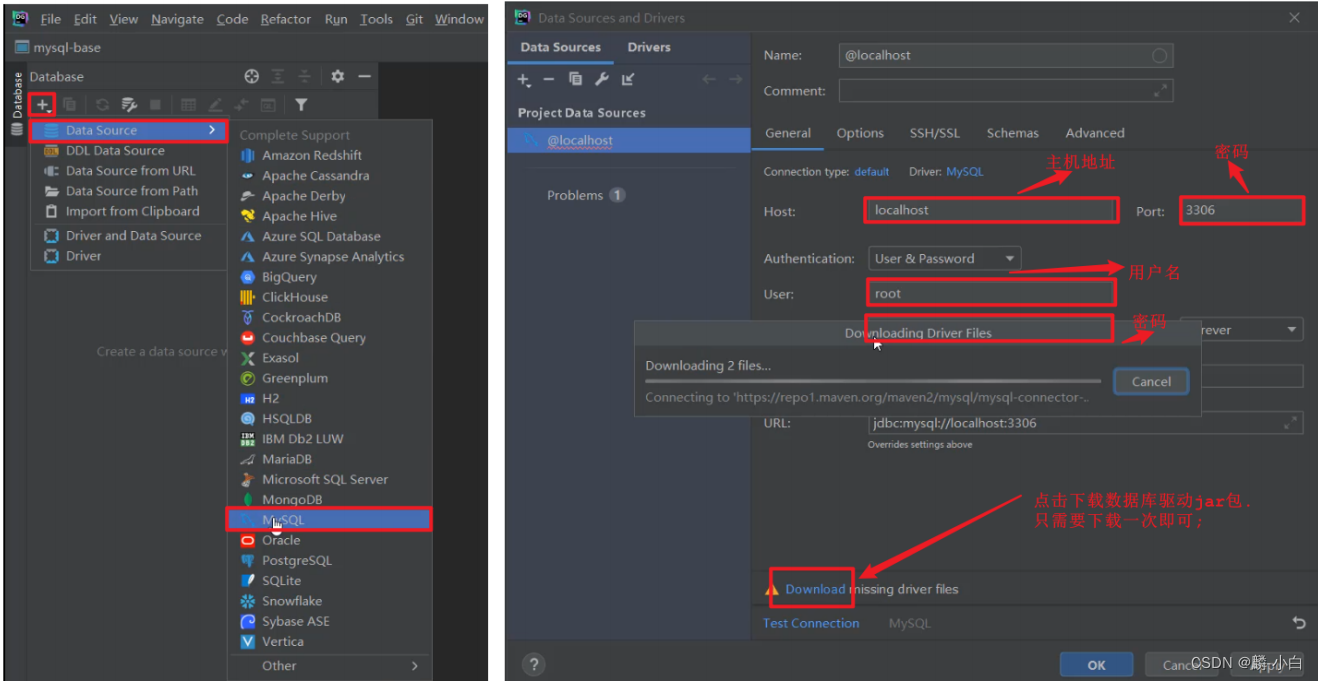
- 配置以及驱动jar包下载完毕之后,就可以点击 "Test Connection" 就可以测试,是否可以连接MySQL,如果出现 "Successed",就表名连接成功了 。
展示所有数据库
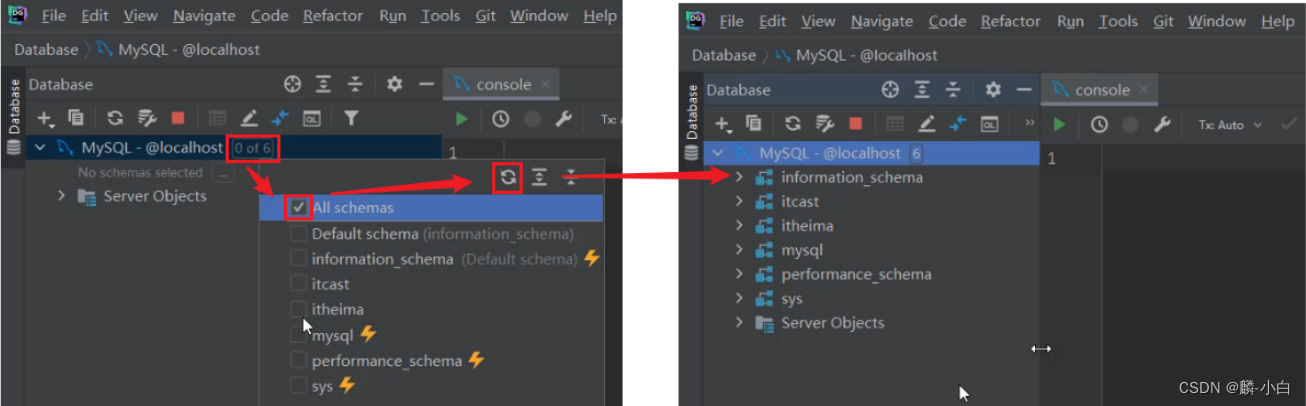
- 连接上了MySQL服务之后,并未展示出所有的数据库,此时,我们需要设置,展示所有的数据库,具体操作如下:
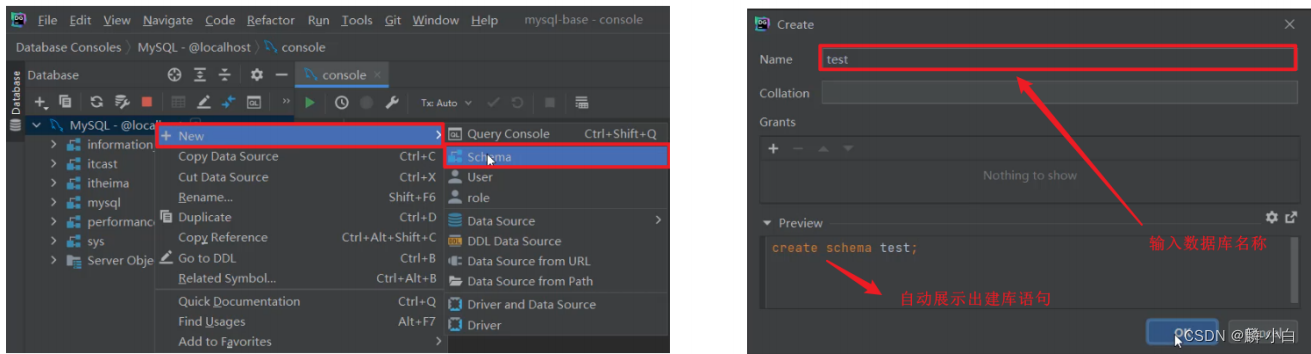
创建数据库
注意:
- 以下两种方式都可以创建数据库:
- create database db01;
- create schema db01;
创建表
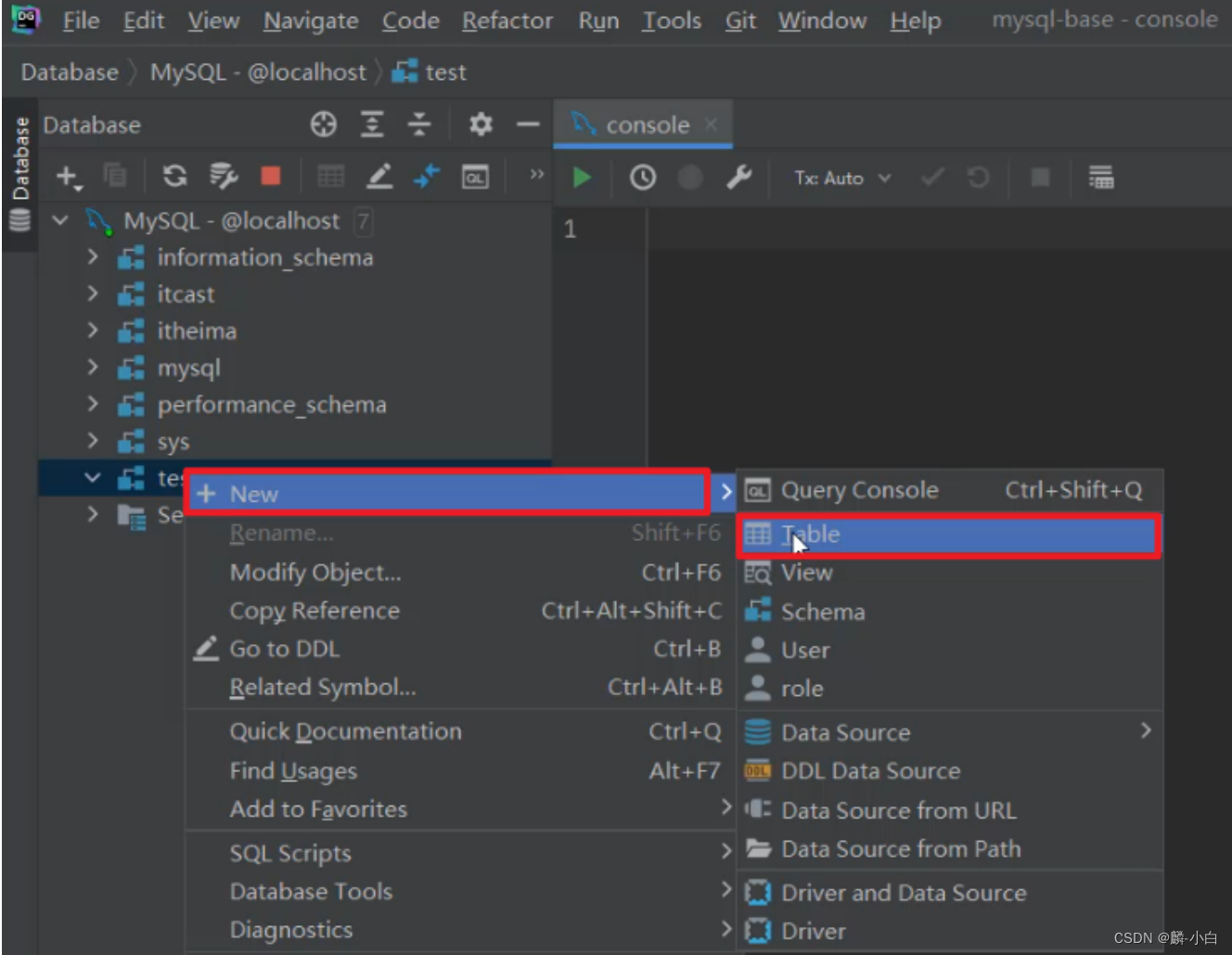
- 在指定的数据库上面右键,选择new --> Table
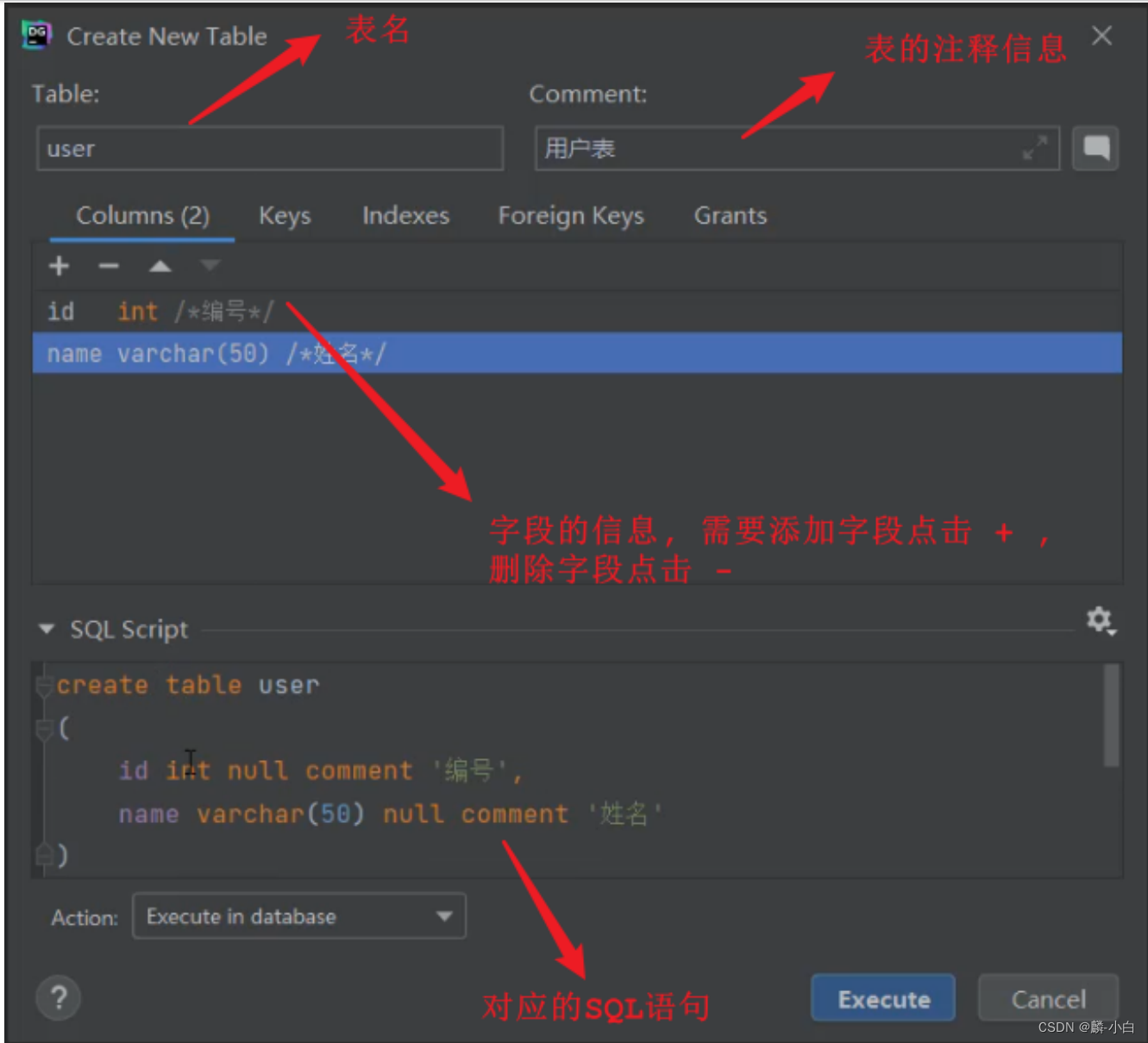
修改表结构
- 在需要修改的表上,右键选择 "Modify Table..."
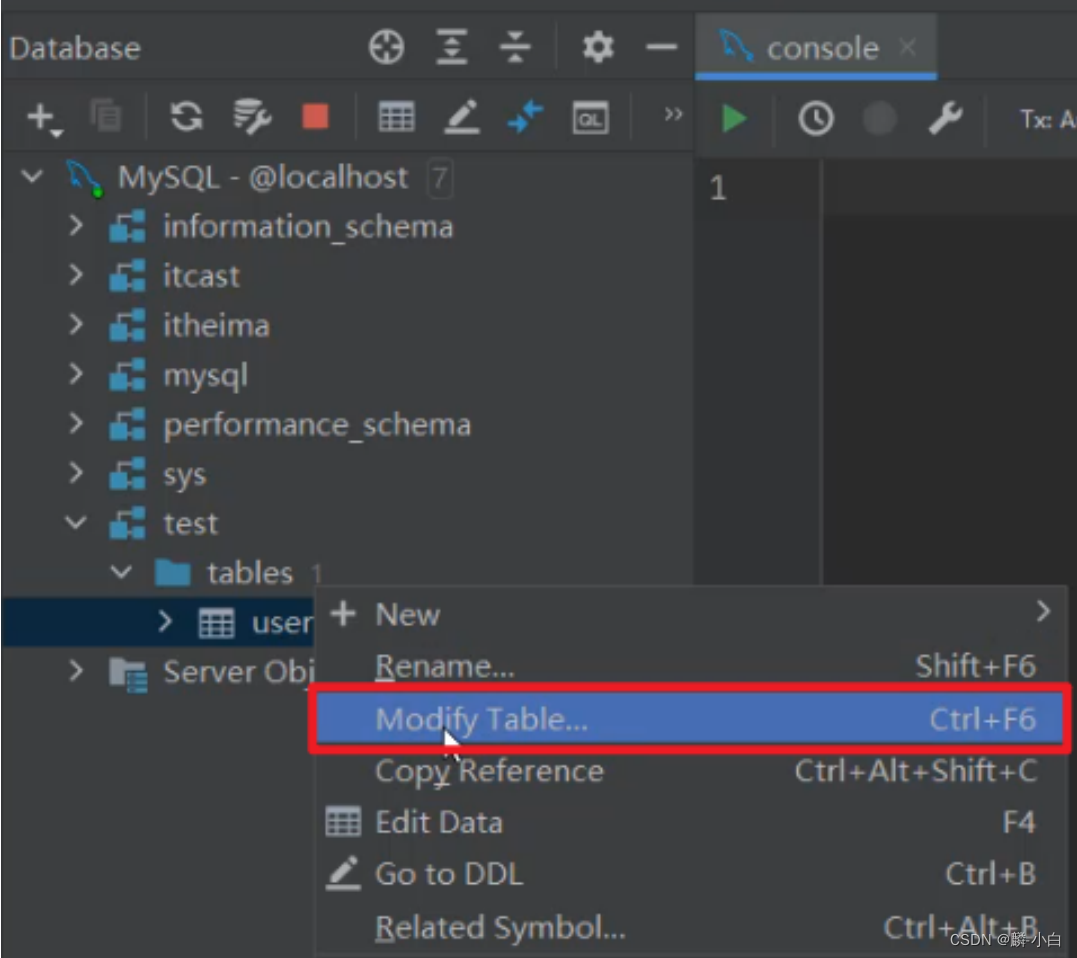
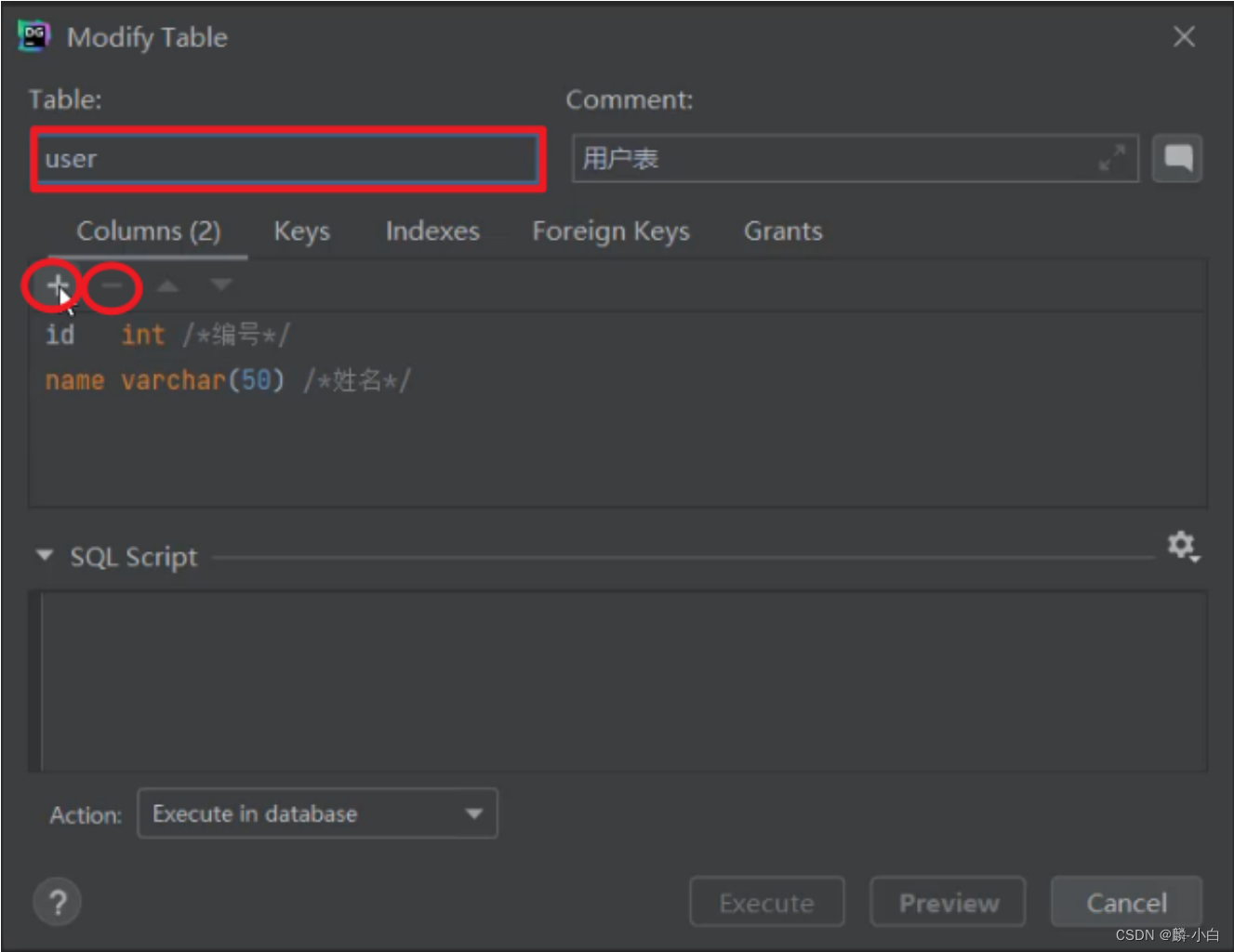
- 如果想增加字段,直接点击+号,录入字段信息,然后点击Execute即可。
- 如果想删除字段,直接点击-号,就可以删除字段,然后点击Execute即可。
- 如果想修改字段,双击对应的字段,修改字段信息,然后点击Execute即可。
- 如果要修改表名,或表的注释,直接在输入框修改,然后点击Execute即可。
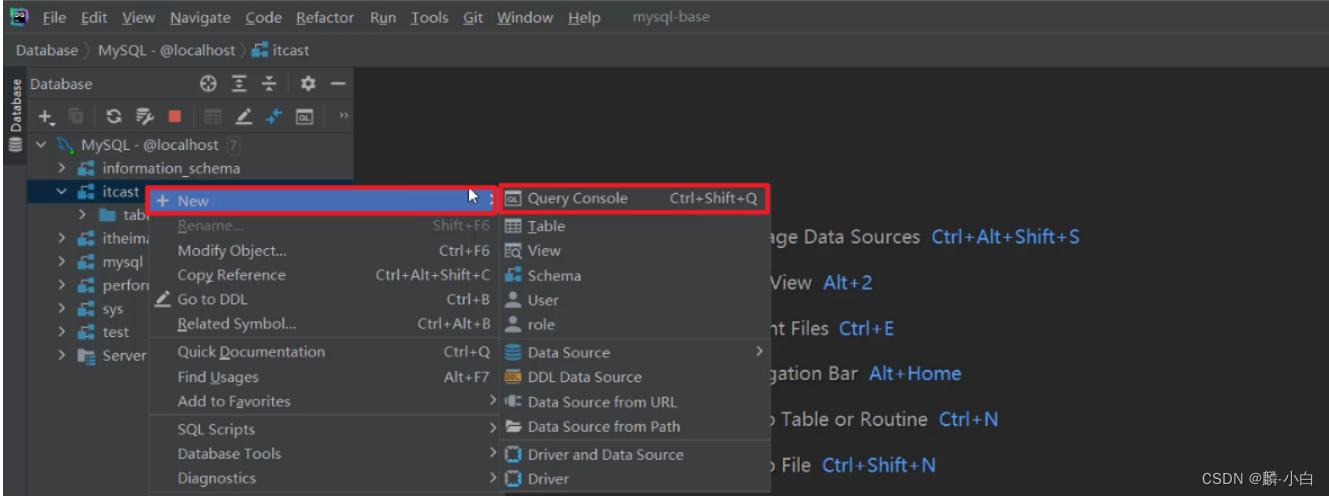
在DataGrip中执行SQL语句
- 在指定的数据库上,右键,选择 New --> Query Console
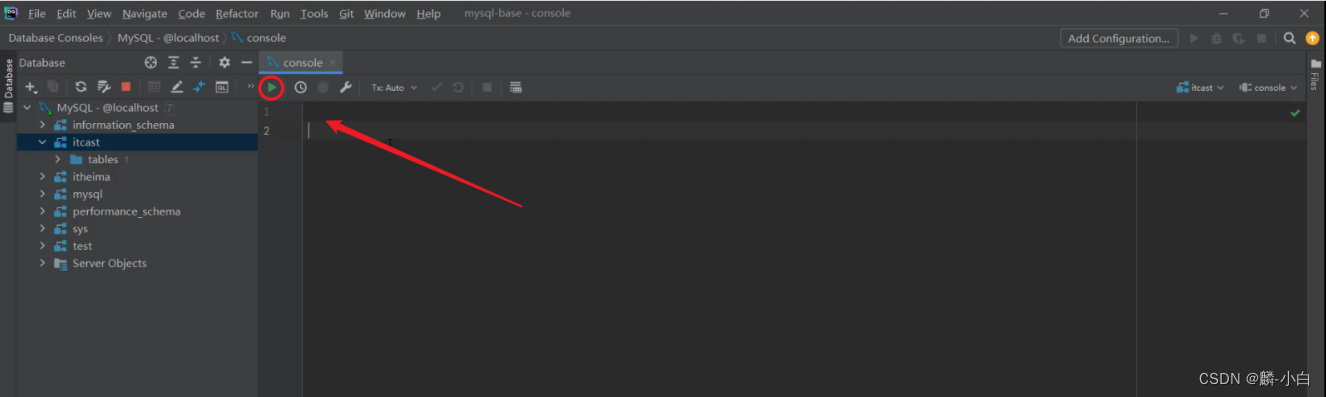
- 然后就可以在打开的Query Console控制台,并在控制台中编写SQL,执行SQL。
![]()
2. DML
2.1 添加数据
- DML英文全称是Data Manipulation Language(数据操作语言),用来对数据库中表的数据记录进行增、删、改操作。
- 添加数据(INSERT)
- 修改数据(UPDATE)
- 删除数据(DELETE)
给指定字段添加数据INSERT INTO 表名 (字段名1, 字段名2, ...) VALUES (值1, 值2, ...); 1
- 案例: 给employee表所有的字段添加数据 ;
insert into employee(id,workno,name,gender,age,idcard,entrydate) values(1,'1','Itcast','男',10,'123456789012345678','2000-01-01');
- 插入数据完成之后,我们有两种方式,查询数据库的数据:
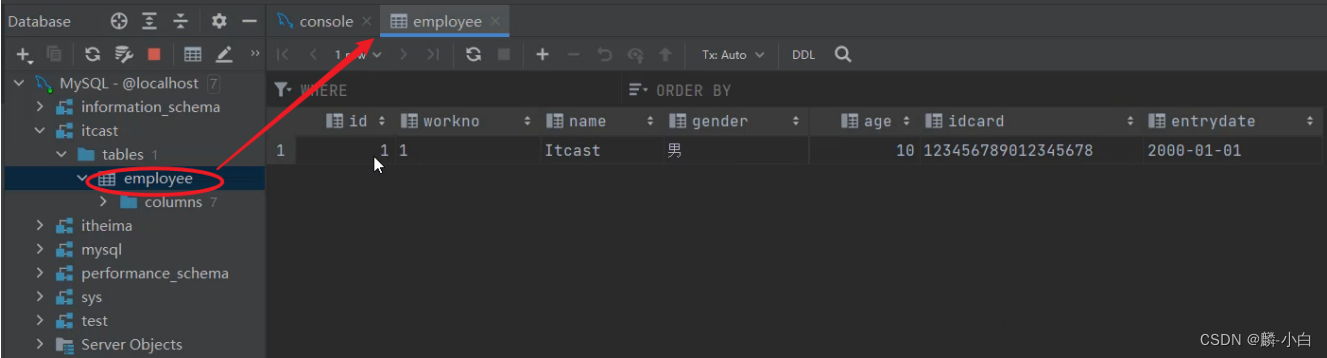
方式一
- 在左侧的表名上双击,就可以查看这张表的数据。
 方式二
方式二
- 可以直接一条查询数据的SQL语句, 语句如下:
select * from employee;案例: 给employee表所有的字段添加数据
- 执行如下SQL,添加的年龄字段值为-1。
insert into employee(id,workno,name,gender,age,idcard,entrydate) values(1,'1','Itcast','男',-1,'123456789012345678','2000-01-01');
- 执行上述的SQL语句时,报错了,具体的错误信息如下:

- 因为 employee 表的age字段类型为 tinyint,而且还是无符号的 unsigned ,所以取值只能在0-255 之间。
 给全部字段添加数据
给全部字段添加数据INSERT INTO 表名 VALUES (值1, 值2, ...);
- 案例:插入数据到employee表,具体的SQL如下:
insert into employee values(2,'2','张无忌','男',18,'123456789012345670','2005-01- 01');批量添加数据INSERT INTO 表名 (字段名1, 字段名2, ...) VALUES (值1, 值2, ...), (值1, 值2, ...), (值 1, 值2, ...) ; INSERT INTO 表名 VALUES (值1, 值2, ...), (值1, 值2, ...), (值1, 值2, ...) ;
- 案例:批量插入数据到employee表,具体的SQL如下:
insert into employee values(3,'3','韦一笑','男',38,'123456789012345670','2005-01- 01'),(4,'4','赵敏','女',18,'123456789012345670','2005-01-01');注意事项:
- 插入数据时,指定的字段顺序需要与值的顺序是一一对应的。
- 字符串和日期型数据应该包含在引号中。
- 插入的数据大小,应该在字段的规定范围内。
2.2 修改数据
- 修改数据的具体语法为:
UPDATE 表名 SET 字段名1 = 值1 , 字段名2 = 值2 , .... [ WHERE 条件 ] ;案例:
- 修改id为1的数据,将name修改为itheima
update employee set name = 'itheima' where id = 1;
- 修改id为1的数据, 将name修改为小昭, gender修改为 女
update employee set name = '小昭' , gender = '女' where id = 1;
- 将所有的员工入职日期修改为 2008-01-01
update employee set entrydate = '2008-01-01';注意事项:
- 修改语句的条件可以有,也可以没有,如果没有条件,则会修改整张表的所有数据。
2.3 删除数据
- 删除数据的具体语法为:
DELETE FROM 表名 [ WHERE 条件 ] ;案例:
- 删除gender为女的员工
delete from employee where gender = '女';
- 删除所有员工
delete from employee;注意事项:
- DELETE 语句的条件可以有,也可以没有,如果没有条件,则会删除整张表的所有数据。
- DELETE 语句不能删除某一个字段的值(可以使用UPDATE,将该字段值置为NULL即可)。
- 当进行删除全部数据操作时,datagrip会提示我们,询问是否确认删除,我们直接点击Execute即可。
![]()
结语
本人会持续更新文章的哦!希望大家一键三连,你们的鼓励就是作者不断更新的动力